基本简介
一、16SrRNA
16SrRNA为核糖体的RNA的一个亚基,16SrDNA就是编码该亚基的基因。细菌rRNA(核糖体RNA)按沉降系数分为3种,分别为5S、16S和23S rRNA。16S rDNA是细菌染色体上编码 rRNA相对应的DNA序列,存在于所有细菌染色体基因中。该序列包含9个高变区和10个保守区,通过对某一段高变区序列(V4区或V3-V4区)进行PCR扩增后进行测序,得到1500bp左右的序列。对于16S测序而言,任何一个高变区或几个高变区,尽管变异性再高,对于某些物种来说,这些高变区也可能十分相近,而能够区分它们的特异性序列片段有可能不在我们的扩增区域内。换言之,非全长的可变区序列覆盖范围不够导至无法鉴定到种。
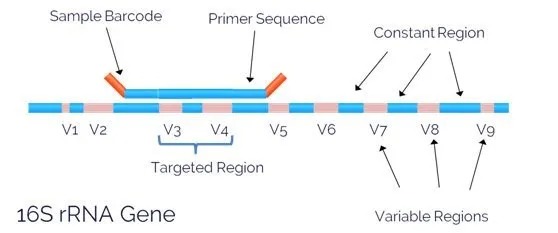
目前来说,16S比较可靠的是用来做菌群的群落分析,物种的组成,多样性分析等,但是由于16S测序本身的性质,想要注释到种水平目前准确性还有待商榷。由于16s是以菌为主体进行研究,想要研究具体的功能目前来说还比较困难。
2、宏基因组
宏基因组研究以环境中所有微生物基因组为研究对象,通过对环境样品中的全基因组DNA进行高通量测序,获得单个样品的饱和数据量,基于denovo组装进行微生物群落结构多样性,微生物群体基因组成及功能,特定环境相关的代谢通路等分析,从而进一步发掘和研究具有应用价值的基因及环境中微生物群落内部、微生物与环境间的相互关系。构建的环境微生物基因集,可为环境中微生物的研究、开发和利用提供基因资源库。
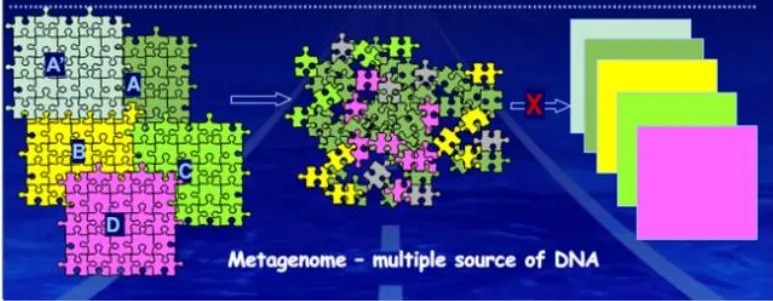
宏基因组测序又能做什么分析呢,首先16s能做的宏基因组都能做,有些还能做的更好,比如宏基因组就可以准确的在种水平上进行相应的注释。除此之外,由于宏基因组可以组装到比对到基因上,那么就可以基于基因水平进行更多的分析,如GO,KEGG功能分析,代谢相关关联分析,疾病关联分析等。对于菌群在疾病的发生发展的解释会更加的细致具体。
总结
如果你有一些临床样本(口腔,鼻腔或粪便等),想了解研究菌群与疾病的关联,那么我们该选16S测序还是宏基因组测序呢? 首先就是研究经费得够,目前来说16S一个只要几百块钱,但是宏基因组测序一个样本需要3、4千,如果经费不够那就选择16S啦。其次和我们的研究目标是密切相关的,假设我们研究的是疾病与对照组间菌群直接的差异,那么16S测序完全够用,而且目前来说除了种水平外,其它的多个水平同样的样本16s注释的物种会更加丰富。当然如果需要研究关键的功能和基因,那么直接选择宏基因组测序即可。














